9月9日,《自然・通讯》(Nature Communications)在线发表了武汉大学药学院孙宇辉教授和张郑宇特聘研究员课题组关于活性天然产物曲张链丝菌素(Streptovaricins)中亚甲基双氧桥结构生物合成机制的重要研究进展[1],博士研究生孙果为本论文第一作者。此研究得到国家重点研发计划、中国科学技术协会青年精英科学家资助计划、微生物代谢国家重点实验室开放课题的大力支持。
抗生素的发现是人类抵抗疾病漫长过程中的关键转折点,但随着在临床上的大量使用,抗生素耐药性已经成为药物研发的重要掣肘。金黄色葡萄球菌是临床上引起感染的常见病原菌,然而,耐甲氧西林金黄色葡萄球菌(MRSA)等耐药菌的出现,使得对这一病原菌感染的治疗变得尤为棘手。因此,发现和创新这类抗耐药菌药物成为了人们的迫切愿望。
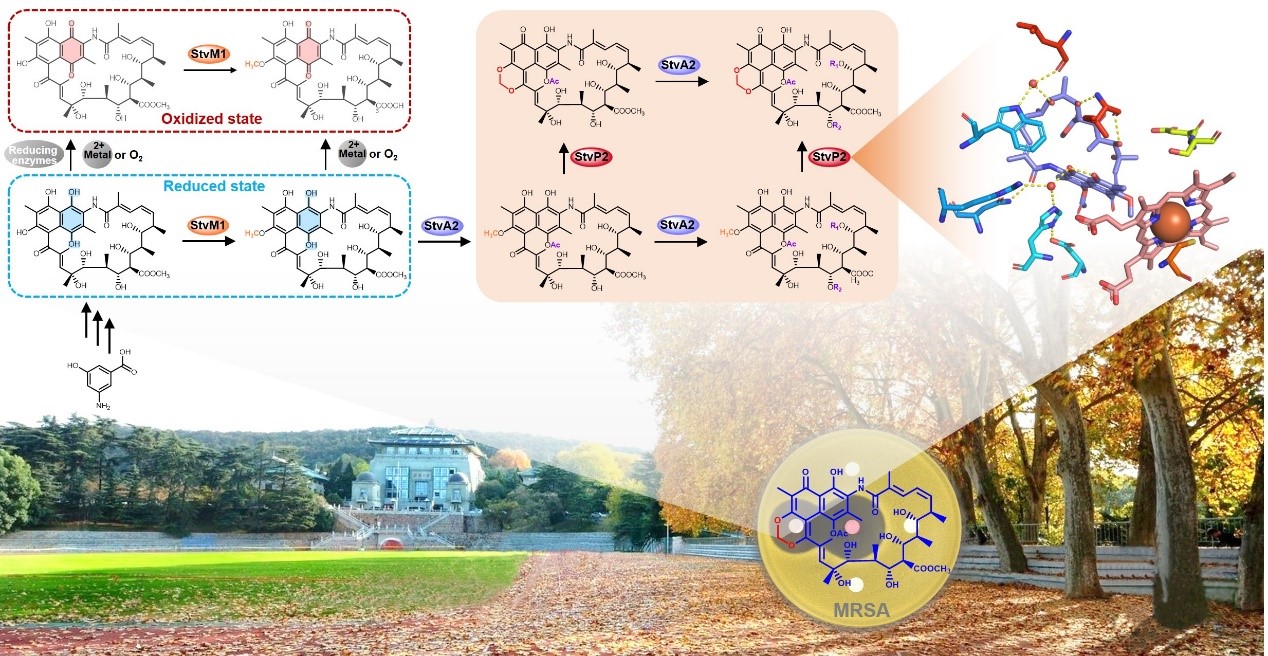
孙宇辉课题组在前期针对抗MRSA活性天然产物的大量研究中,从武汉大学校园土壤中分离到的一株链霉菌(Streptomyces spectabilisCCTCC M2017417)中发现了一类具有抗MRSA活性的萘安莎类抗生素,经鉴定为曲张链丝菌素,它在结构上与著名的肺结核明星药治疗物利福霉素同属一个家族。通过微生物分子遗传学、生物化学等方法,对曲张链丝菌素生物合成中参与活性结构的多个细胞色素P450酶编码基因进行了全面探索,并对定向遗传改造获得的曲张链丝菌素系列新结构衍生物的抗MRSA活性进行了表征。相关的研究成果已发表于《美国化学会・化学生物学》(ACS Chemical Biology, 2017, 12: 2589-2597)[2]。在此基础上,近期结合结构生物学手段,对曲张链丝菌素中独特且与抗MRSA活性密切偶联的亚甲基双氧桥结构的生物合成机制进行了深入的揭示。
首先,根据生物信息学分析及前期实验结果,推测该菌细胞色素P450酶StvP2有可能是负责曲张链丝菌素结构中亚甲基双氧桥结构形成的关键酶。从其突变株中分离获得的关键中间体及相应的体外酶学催化结果充分地证实了这一推测。随后,通过多重遗传敲除所构建的突变株中系列中间体的分离和鉴定,对该酶催化特性进行了探究,表征出它所催化的亚甲基双氧桥合成反应的非典型底物抑制动力学特征。
为了从本质上解析StvP2催化亚甲基双氧桥结构的形成机制,通过与张郑宇特聘研究员课题组合作,利用X-ray晶体衍射分别解析了该酶在无底物和有底物结合时的蛋白晶体结构,揭示了其中独特的催化三联体所负责的“酮式-烯醇式”互变异构,并通过单加氧反应及后续的成环过程,最终形成具有亚甲基双氧桥结构的终产物,该催化机制进一步被关键氨基酸残基系列点突变及生化证据所证实。
为完整阐释亚甲基双氧桥形成的生物合成途径,巧妙地借用了菠菜来源的电子传递和还原系统,在体外成功地重建了由甲基化酶StvM1、酰基化酶StvA2和氧化酶StvP2共同参与的亚甲基双氧桥结构形成过程。这一研究进展向我们展示了曲张链丝菌素及其该类化合物生物合成过程中的核心步骤,深入解析了亚甲基双氧桥形成的酶学机制,为后续曲张链丝菌素类化合物的理性结构改造及其抗MRSA活性药物的研发提供了精准的“导航图”。
孙宇辉教授和张郑宇特聘研究员同属药学院邓子新院士团队成员,均为近年来从海外引进人才,他们分别聚焦于微生物遗传学和结构生物学领域,此项工作从学科的互补性和交叉性充分体现了团队合作在解答复杂科学问题的强大优势。
相关论文链接:
[1] Guo Sun, Chaoqun Hu, Qing Mei, Minghe Luo, Xu Chen, Zhengyuan Li, Yuanzhen Liu, Zixin Deng, Zhengyu Zhang* and Yuhui Sun*, Uncovering the cytochrome P450-catalyzed methylenedioxy bridge formation in streptovaricins biosynthesis. Nature Communications 2020, 11: 4501. [https://www.nature.com/articles/s41467-020-18336-5]
[2] Yuanzhen Liu, Xu Chen, Zhengyuan Li, Wei Xu, Weixin Tao, Jie Wu, Jian Yang, Zixin Deng and Yuhui Sun*, Functional analysis of cytochrome P450s involved in streptovaricin biosynthesis and generation of anti-MRSA analogues. ACS Chemical Biology 2017, 12: 2589-2597. [https://pubs.acs.org/doi/10.1021/acschembio.7b00467]